La mejora genética clásica tiene limitaciones para abordar los caracteres relacionados con parámetros de calidad de carne debido a distintos factores, como su registro costoso y tardío o los antagonismos de estos caracteres con los de eficiencia y rendimiento. En este contexto, las tecnologías de análisis genético-molecular pueden permitir el diseño de programas de mejora más precisos y eficaces, basados en el empleo de marcadores genéticos.
Los estudios moleculares de genes candidatos, cuyas funciones biológicas pueden relacionarse directamente con caracteres de interés, han permitido identificar algunos marcadores genéticos (variaciones puntuales en el genoma) con efectos relevantes, aunque en muy pocos casos se consideran mutaciones causales de los efectos observados. Entre ellas se pueden resaltar los polimorfismos encontrados en el gen LEPR, relacionado con el control del balance energético, el apetito y el engrasamiento; el gen SCD, implicado en la síntesis y acumulación de grasa monoinsaturada (ácido oleico principalmente); o el gen PCK1, relacionado con el contenido de grasa intramuscular y la calidad de la carne. Por otra parte, las herramientas de secuenciación masiva y de genotipado masivo de marcadores de ADN permiten realizar distintos tipos de análisis de asociación del genoma con los caracteres de interés socioeconómico que contribuyen a la comprensión de su base molecular en las especies domésticas. Estas herramientas y su combinación se han empleado para la identificación de regiones genómicas y potenciales mutaciones causales para distintos aspectos productivos de crecimiento, rendimiento, composición tisular y calidad de carne en cerdos ibéricos. Los resultados resaltan el interés de marcadores localizados en genes conocidos relacionados con el metabolismo lipídico, como ACACA, ELOVL6 o SCD, proteólisis, como CAPN1; o la regulación de la homeostasis de la energía, como FTO o PRKAG3, así como otros marcadores localizados en muchos otros genes de función menos clara.
Para algunos genes y caracteres citados, las asociaciones del genoma se han contrastado ya en distintos entornos genéticos y los marcadores están hoy día en disposición de ser empleados en la práctica de los programas de mejora. En relación con los resultados más recientes, las asociaciones deben ser validadas suficientemente en distintas poblaciones comerciales antes de su aplicación definitiva. Estos marcadores permitirán la incorporación de la información molecular en los programas de mejora, junto a la siempre necesaria información tradicional productiva y genealógica, mejorando la precisión de las estimas de los valores mejorantes o empleándose para descartar reproductores con un genotipo desfavorable. Por otra parte, la selección genómica es una novedosa estrategia alternativa, basada principalmente en el uso de un gran volumen de información molecular, que podría en el futuro mejorar la precisión de la selección y acelerar la mejora, reduciendo la necesidad de registrar datos fenotípicos.
La mejora genética clásica y sus limitaciones
La mejora genética clásica, que no está basada en marcadores, ha sido muy importante para aumentar la eficiencia económica en todas las especies domésticas. Sus efectos son permanentes, acumulativos y normalmente muy rentables, permitiendo tasas anuales de progreso genético de considerable magnitud. Este tipo de mejora se basa en el uso de la información fenotípica (por ejemplo los registros productivos) y la información genealógica. Estos datos, no moleculares, permiten aplicar el denominado modelo animal y calcular el parámetro fundamental para la selección, el valor mejorante de los animales (EBV, Estimated Breeding Value) mediante la metodología estadística BLUP (Best Linear Unbiased Prediction). Estos valores mejorantes nos permiten elegir como reproductores a los animales que son genéticamente mejores, elevando de esta forma el nivel genético de toda la población. Este tipo de mejora ha sido especialmente eficaz en razas cosmopolitas para la mejora de caracteres relacionados con el rendimiento magro o la prolificidad y también se aplica con éxito en diversas razas autóctonas de los países más desarrollados.
A diferencia de las razas magras cosmopolitas, las experiencias de mejora genética en cerdo ibérico comenzaron muy tarde, en los primeros años de la década de los 90 del siglo pasado, y no hay demasiados estudios en los que se analice la implementación de estos programas. Este hecho puede estar relacionado con la estructura tradicional de la población y con la especificidad del sistema de producción en extensivo. Desde el punto de vista de la evaluación genética y la selección, las primeras iniciativas se llevaron a cabo por el grupo de porcino del Departamento de Mejora Genética de INIA, cuyas experiencias se trasladaron al Programa Oficial de Mejora de la Raza, impulsado desde el Ministerio de Agricultura, Pesca y Alimentación de la época con la publicación de los reglamentos del esquema de valoración genética para la raza porcina Ibérica (1992 y 2011). AECERIBER ha sido desde el inicio la asociación responsable de la gestión del Libro Genealógico y del Programa de Selección oficiales de la raza.
El diseño inicial de este programa buscaba un doble objetivo. Por un lado, conseguir una mejora de los caracteres de crecimiento registrando pesos a edades tempranas y desarrollar pruebas de ciclo completo realizadas en una misma finca de montanera (AECERIBER, 1998). Posteriormente, se incluyó el porcentaje de grasa intramuscular e incluso el perfil de ácidos grasos como caracteres de calidad. En un principio los caracteres reproductivos (tamaños de camada, aptitud materna) fueron excluidos en el programa debido a su baja heredabilidad, el tamaño pequeño de las explotaciones y la falta de conexiones genealógicas (Silió, 2000), pero actualmente, el programa oficial sí incluye estos caracteres. Actualmente el programa de mejora, que está centrado principalmente en caracteres de crecimiento y reproductivos, está implementado de forma exitosa y se ejecuta en general mediante colaboraciones privadas y público-privadas.
Sin embargo, los métodos de selección genética clásica tienen limitacio nes especialmente para abordar la mejora de caracteres relacionados con paráme tros de calidad de carne debido a distintos factores, como su registro costoso y tardío o los antagonismos de estos caracteres con los de eficiencia y rendimiento. Por ejemplo, existe una correlación negativa entre los porcentajes de lomos y jamones respecto a la canal y el contenido de grasa intramuscular, de modo que la selección para rendimiento de piezas puede afectar negativamente a la calidad y viceversa. Puede haber también respuestas correlacionadas no deseadas en relación a la composición de ácidos grasos. La mejora del rendimiento de jamones, paletas y lomos puede aumentar el contenido en ácido linoleico, afectando a la consistencia de la grasa y a aspectos de calidad organoléptica de los productos curados. Por otra parte, cuando se evalúan hermanos de camada sin datos productivos propios, sus valores mejorantes estimados por BLUP son idénticos dado que se basan en la información fenotípica de sus parientes, que son los mismos, y en esta situación no podemos discriminar cuál de los hermanos es mejor para utilizarlo como reproductor.
En este contexto, los avances en las tecnologías de análisis genético molecular pueden permitir el diseño de programas de mejora más precisos y eficaces, basados en el empleo de marcadores genéticos.
Información molecular. Marcadores genéticos
Tradicionalmente, la mejora genética ha utilizado los conocimientos en genética cuantitativa para predecir los animales con una composición genética más adecuada a los objetivos de selección. Pero las últimas décadas han traído un desarrollo enorme de todas las técnicas de análisis del genoma a distintos niveles biológicos, y estas técnicas moleculares están permitiendo el análisis de los genes y la biología celular de forma cada vez más exhaustiva y eficaz, permitiendo el estudio de marcadores moleculares de forma masiva. Por ello, el desarrollo de la genética molecular ha revolucionado los planteamientos de la mejora genética clásica ya que proporciona información directa acerca del patrimonio genético de los individuos.
El genoma de mamíferos está compuesto por 3.000 millones de nucleótidos (unidades que componen el ADN) y el 99,9% del genoma es idéntico entre individuos de la misma especie. Esto significa que el efecto de la constitución genética sobre el fenotipo se debe a ese O,1% variable en la población, responsable de que unos individuos tengan mejores o peores atributos. Las variaciones o polimorfismos en la secuencia de ADN es lo que conocemos como marcadores genéticos o marcadores moleculares, que pueden determinar la variabilidad fenotípica. Los microsatélites, secuencias repetidas de un fragmento de ADN, han sido los marcadores más conocidos y fueron muy prácticos para estudios de poblaciones o para verificaciones de paternidad. Actualmente, el marcador más utilizado, simple y abundante es el SNP (Single Nucleotide Polymorphism), que es un cambio de un solo nucleótido en la secuencia de ADN (Figura 1). Es un marcador fácil de genotipar y existen muchas alternativas tecnológicas que permiten trabajar bien con SNPs individuales o bien con una cantidad enorme de ellos.
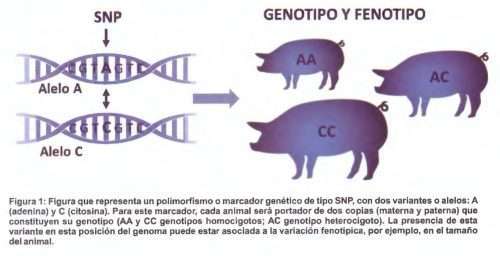
Podemos clasificar los marcadores moleculares en 2 tipos. En 1º lugar, los marcadores que llamamos causales o funcionales son aquellas variantes del ADN que influyen directamente en el metabolismo del animal y de este modo afectan al fenotipo. Son marcadores que están normalmente dentro de genes funcionalmente relacionados con un carácter. En 2º lugar, los marcadores neutros son variantes del ADN que en principio no tienen ningún efecto sobre el fenotipo, pero sirven para rastrear cómo se heredan las distintas regiones del genoma que sí pueden tener efecto directo sobre el fenotipo. Estos marcadores neutros son los que están contenidos en los denominados chips de genotípado de SNPs desarrollados por diversas empresas de genética que permiten analizar simultáneamente decenas o centenas de miles de SNPs. En el caso del ganado porcino, los chips de genotipado más usados son los de la casa comercial Ilumina que tiene alrededor de 60.000 SNPs (SNP60 Genotyping BeadChip) y el comercializado por Neogen con más de 70.000 (GGP-Porcine HD), aunque existe también una plataforma de alta densidad que permite analizar cerca de 700.000 SNPs simultáneamente (Axiom Porcine Genotyping Array de Affimetrix). Este tipo de SNPs permiten relacionar segmentos específicos del ADN con diferencias productivas para determinados caracteres.
Para el empleo de información molecular en la mejora del ganado es necesario por tanto encontrar y analizar cuáles de esas variantes o bien· qué regiones genómicas son las que determinan las diferencias fenotípicas entre individuos en caracteres de interés. Para ello, es necesario en 1º lugar descubrir esas variantes, en 2º lugar, determinar cuáles son interesantes y en 3º lugar integrar el análisis en los programas de mejora.
Estrategias de estudio
Existen 2 estrategias para determinar qué variantes se asocian a los caracteres fenotípicos de interés que queremos potenciar.
1. Estudio de gen candidato
Este enfoque consiste en centralizar el estudio en uno o unos pocos genes de función biológica conocida y relaciona da con el carácter de interés. Lo más habitual son los estudios estructurales, que consisten en la búsqueda de variantes genéticas de esos genes, generalmente mediante secuenciación del ADN, seguido del estudio estadístico para asociar la variación en el ADN con la variación del carácter. Estos estudios pueden complementarse con estudios funcionales, en los que se evalúa la expresión o función del gen en un determinado momento y tejido (músculo, grasa, hígado, etc.) para inferir su relación con un fenotipo.
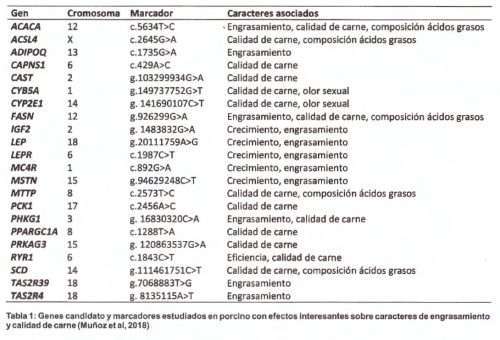
Los estudios moleculares de genes candidato han permitido identificar algunas mutaciones con efectos relevantes. En cerdos ibéricos estos estudios se han centrado principalmente en genes relacionados con el control del balance energético (como LEP, LEPR o MC4R), de la síntesis de lípidos (como FASN, ACACA o SCO) y de la proteólisis (calpainas y calpastatinas).
Se explican a continuación los efectos de algunos de estos marcadores en genes candidato, especialmente interesantes para mejora o que han sido objeto de numerosos estudios científicos.
El gen LEPR codifica para el receptor de leptina, receptor que se expresa en el hipotálamo, donde regula el balance energético y el consumo de alimentos. Nuestro grupo de investigación ha analizado este gen en profundidad en distintas poblaciones porcinas y ha caracterizado los efectos de un SNP que se asocia de modo consistente a caracteres relacionados con balance energético (Ovilo et al, 2005, 2010). El alelo T, fijado en ibérico (es decir, tiene una frecuencia 100%, todos los animales tienen genotipo TT), aumenta el apetito y el engrasamiento, incluyendo el tocino dorsal además de la grasa intramuscular, por lo que se ha relacionado también con otros caracteres de calidad de carne como la terneza. Dado que este marcador tiene el alelo favorable fijado en ibérico, puede ser útil en la selección de machos Duroc empleados en la producción 50%, en caso de que interese aumentar el engrasamiento general del animal.
Otro gen candidato con relevancia para la calidad es el gen SCD que codifica para la enzima delta 9 desaturasa. Este gen codifica el enzima necesario para la síntesis de ácidos grasos oleico y palmitoleico por lo que es muy importante en la calidad de los productos. En este gen se han caracterizado 3 SNPs, localizados en zonas reguladoras importantes y que se asocian con el contenido en oleico en distintos tejidos (Estany et al, 2014) y en algunos de los trabajos también se asocia con el contenido de grasa intramuscular (Fernandez et al, 2017). La combinación de alelos favorable (C-T-A) está a alta frecuencia en ibérico (98%, Muñoz et al, 2018), por lo que hay poco margen de mejora asociada a este SNP en poblaciones ibéricas puras, pero podría utilizarse en Duroc para la mejora de la mono insaturación de carne y grasa e infiltración grasa en músculo. Además, resultados recientes, todavía sin publicar, de nuestro grupo muestra un efecto de este marcador sobre caracteres de crecimiento y eficiencia.
El gen PCK1 codifica una enzima que está en una intersección de rutas metabólicas clave que repercuten en la producción de glúcidos y lípidos. En este gen se ha encontrado un polimorfismo interesante que tiene efecto en la distribución de la grasa y calidad de carne (Latorre et al, 2016). El polimorfismo tiene efectos opuestos sobre el espesor del tocino dorsal y sobre el porcentaje de grasa intramuscular, lo que facilitaría la selección independiente de estos 2 caracteres, a pesar de su correlación genética positiva. El alelo A está asociado con un mayor contenido de grasa intra muscular y también con una mayor capacidad de retención de agua, por lo que es un marcador de gran interés para la mejora de la calidad de carne. Como en el ejemplo anterior, el ganado ibérico presenta este alelo favorable a frecuencia elevada (96%, Muñoz et al, 2018), por lo que su utilidad se limita a la eliminación del alelo desfavorable de la población ibérica o bien al empleo en mejora de otras razas.
Un trabajo reciente de los investigadores del Centro de INIA-CSIC en Zafra ha detectado asociaciones interesantes de distintos genes con caracteres de calidad de carne en una población ibérica (Fernandez-Barroso et al, 2020). En este caso, los hallazgos permiten proponer marcadores que serían de utilidad en poblaciones ibéricas puras, puesto que son marcadores que presentan segregación en la raza, es decir, hay suficientes individuos con diversidad genética para ese SNP. Los resultados más interesantes son los relativos a la asociación del gen PRKAG3 con el color de la carne y de los genes PRKAG3, MYH3, CASP3, CAPN1 y CTSL con distintos caracteres de textura y pérdida de agua. Por último, en este apartado, hemos obtenido en una ganadería comercial resultados interesantes más recientes, pendientes de publicación, en caracteres como el porcentaje de grasa intramuscular, el perfil de ácidos grasos o el porcentaje de piezas nobles en la canal.
2. Estudios de barrido genómico o asociación de genoma completo (GWAS)
Los estudios de barrido genómico se basan en el empleo de paneles amplios de marcadores neutros repartidos homogéneamente a lo largo de todo el genoma. Por ello, para su implementación ha sido decisivo el desarrollo de los denominados chips de genotipado masivo comercial/es. Los estudios de asociación del genoma, conocidos como GWAS (Genome-Wide Association Studies), tienen el interés
científico de localizar las regiones del genoma relacionadas con un carácter. En estos trabajos se analizan un número importante de animales (cientos o miles) con un chip de genotipado masivo y se miden los caracteres fenotípicos de interés. Después se combina la información genotípica y fenotípica en un estudio estadístico complejo que permite identificar las regiones que afectan a cada carácter. El resultado de estos estudios son gráficas denominadas manhattan plots, nombre inspirado en el sky-line de Nueva York (Figura 2), que representan las distintas posiciones del genoma, de modo que cada punto corresponde a un SNP, y su posición en el eje de ordenadas «y» indica la probabilidad de que ese SNP este asociado un carácter en particular.
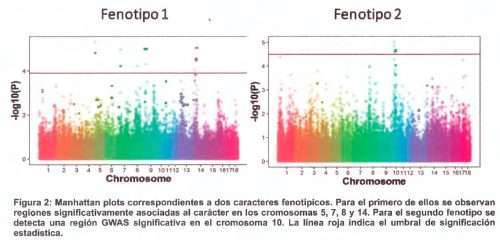
La mayor parte de estudios GWAS se han realizado con poblaciones porcinas experimentales o poblaciones comerciales de distintas razas. En el caso del ibérico, son escasos los estudios GWAS realizados en poblaciones ibéricas puras y poblaciones comerciales (Pena et al, 2019). Los trabajos realizados por nuestro grupo, tanto en poblaciones experimentales como comerciales ibéricas (Martínez Montes et al, 2018; Muñoz et al, 2022) han permitido la identificación de regiones genómicas asociadas a caracteres relevantes para la producción y la calidad de los productos, como el peso al sacrificio, el espesor del tocino dorsal, el contenido de grasa intramuscular, el peso de la maza, el peso del lomo o rendimiento de piezas nobles. En relación a la composición y el metabolismo tisular de ácidos grasos, que son muy relevantes para la calidad organoléptica y nutricional de la carne, también se han detectado regiones significativamente asociadas a parámetros de gran interés como el contenido de ácidos grasos omega 6 o el contenido de ácido linoleico.
La identificación de estas regiones GWAS significativamente asociadas a caracteres de interés, permite identificar nuevos genes candidatos aplicando el criterio de su posición en el mapa genético, coincidente con el pico GWAS, además de su función biológica. Así, se pueden resaltar los genes RXRB, ELOVL6, FTO o FADS1 que se han propuesto, y en algunos casos estudiado, como candidatos biológicos y posiciona les para algunos caracteres relevantes como los de composición de ácidos grasos (Crespo-Piazuelo et al, 2020; Pena et al, 2019; Muñoz et al, 2022). Esto permite el planteamiento de nuevos estudios encaminados a la búsqueda de los polimorfismos específicos responsables de las asociaciones detectadas. Actualmente, nuestro grupo de INIA-CSIC está desarrollando nuevos estudios GWAS en distintas poblaciones comerciales de cerdos Ibéricos puros.
Integración de la información mole cular en programas de mejora
1. Selección asistida por marcadores
Una vez identificadas mutaciones o marcadores que se consideren causales, las podemos utilizar directamente en los programas de mejora asistida por marcadores (MAS, Marker Assisted Selection) dado que son mutaciones con efectos conocidos y dimensionados. Existen distintas alternativas de empleo según a qué frecuencia se encuentren en la población.
Una 1ª opción es utilizar la información del genotipo de genes candidato en combinación con la evaluación BLUP (Fernando & Grossman, 1989). Sin embargo, con relación al engrasamiento y a la calidad de la carne, los cerdos ibéricos suelen tener las variantes favorables a una frecuencia elevada, como se ha comentado previamente para algunos de los ejemplos de genes candidato (LEPR, SCD, o PCK1). Por ello, una 2ª opción en este caso sería descartar los candidatos a reproductores en función de su genotipo, eliminando aquellos que sean portadores de alelos con efectos negativos sobre el carácter de interés, es decir, eliminar los ‘malos’ más que seleccionar los ‘buenos’.
En el caso de las poblaciones cruzadas el margen de mejora suele ser mayor porque existe la posibilidad de mejora por la vía de los machos Duroc. En estas poblaciones Duroc suele observar se segregación de marcadores interesan tes, por lo que se puede seleccionar la variante favorable o bien, en caso de que se encuentre a una frecuencia muy baja que dificulte su selección porque hay muy pocos individuos portadores del «alelo bueno», se pueden implementar diseños enfocados a mejorar la homogeneidad de los caracteres de interés, seleccionando únicamente homocigotos en las razas puras. De este modo se maximiza la frecuencia de heterocigotos en la F1. La presencia en la población cruzada de animales con un solo genotipo posible (heterocigotos) nos va a ayudar a reducir la heterogeneidad de los caracteres de interés, aunque no se maximice su valor.
2. Selección genómica
Los avances en el desarrollo de plataformas de genotipado masivo de SNPs ponen a nuestro alcance una nueva metodología de evaluación genética basada en información molecular, que es la selección genómica. Es importante distinguir esta selección genómica de la MAS. En la MAS los marcadores sirven de complemento a la selección tradicional y al índice BLUP, es decir, se sigue usando además del marcador, la información fenotípica y genealógica. Por el contrario, en la selección genómica los marcadores sustituyen a la información fenotípica. En este método de selección, la disponibilidad de información del genotipo de un volumen muy elevado de SNPs en un volumen muy elevado de animales, permite estimar el valor mejorante o valor genómico de los animales sin conocer su fenotipo (Meuwis sen et al, 2001). Se trata de una selección asistida por marcadores a gran escala, en la que no necesitamos el conocimiento previo de polimorfismos causales, ni necesitamos identificar previamente regiones GWAS. Pero para implementar la selección genómica es necesario conocer cómo se asocia cada uno de los SNPs contenidos en el chip de genotipado con los caracteres que nos interesan. Es decir, necesitamos una población de referencia, numerosa, representativa y conectada a los animales que queremos evaluar, en la que se analiza tanto el genotipo (chip de SNPs) como el fenotipo (datos productivos). En esta población de referencia se establecen las relaciones entre ambos parámetros (genotipo-fenotipo) para poder construir una ecuación de predicción, con la que posteriormente podremos estimar los valores mejorantes en la población de evaluación (GBV, Genomic Breeding Values), sin necesidad de datos fenotípicos, solo con la información de genotipado (Figura 3).
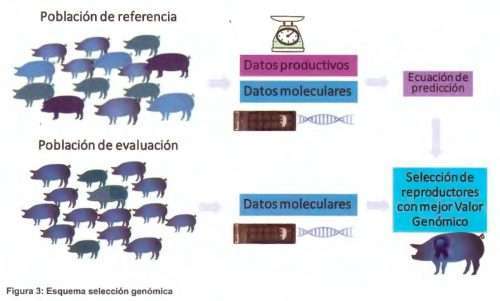
Esta herramienta es muy potente y puede permitir una selección más precisa y temprana y una mejor selección entre hermanos, acelerando en definitiva el progreso genético, pero tiene algunos problemas y limitaciones, que podrían comprometer su empleo en ciertas razas o poblaciones, como el caso del cerdo ibérico. La implantación de un programa de selección genómica requiere una inversión muy grande para generar y analizar la población de referencia, por lo que debe hacerse un análisis de costes y beneficios. Por otra parte, la población de referencia tiene que ser representativa, hay que elegirla cuidadosamente, y debe estar conectada a todas las poblaciones que se quieran evaluar. La mayor parte de investigación y avances en este sistema se han realizado en ganado como el vacuno lechero, en el que hay una estructura piramidal muy clara y hay mucha difusión internacional de la mejora, alcanzando los toros seleccionados elevados precios en el mercado que justifican la inversión realizada. En el caso del ibérico hay varias iniciativas en marcha para empezar a implementarla, aunque probablemente su potencial para cambiar las estructuras de mejora se desarrolle más a largo plazo.
Conclusiones
Con relación a los marcadores disponibles para su uso en mejora, podemos distinguir dos grandes grupos. Por un lado, están aquellos para los que se dispone de información de estudios de asociación en distintas poblaciones e incluso también estudios funcionales. Estos marcadores han mostrado efectos consistentes en los distintos trabajos, en algunos casos considerándose mutaciones causales de los efectos observados, de modo que ya estarían en disposición de ser empleados en programas de mejora. En principio se podría esperar que los genes que afectan a calidad de carne tengan los alelos favorables fijados en ibérico, sin embargo, nuestros resultados recientes (Muñoz et al, 2018) sugieren que, al contrario de lo que se suele asumir, los alelos favorables a la calidad de carne no están fijados del todo, existe variabilidad, lo que justifica la selección para erradicar los alelos desfavorables, especialmente en aquellos con efecto de mayor magnitud e importancia.
Con relación a los resultados de trabajos de asociación más recientes, las asociaciones deben ser validadas suficientemente en distintas poblaciones comerciales antes de su uso práctico. Estos marcadores permitirán la incorporación de la información molecular en los programas de mejora, junto a la información tradicional productiva y genealógica, mejorando la precisión de las estimas de los valores mejorantes o empleándose para descartar reproductores con un genotipo desfavorable.
Por último, la selección genómica ofrece nuevas posibilidades de mejora que podrían ser interesantes y rentables en el ámbito de la producción del cerdo ibérico, pero que requieren aun de la realización de trabajos que evalúen las necesidades organizativas y de estructura para su implementación, así como su potencial rentabilidad para valorar objetivamente su idoneidad en este tipo de producción singular.
Autores
Cristina Óvilo, María Muñoz, Juan María Garcia-Casco. Departamento Mejora Genética Animal, INIA-CSIC, Madrid.



Deja un comentario