INTRODUCCIÓN
La variedad de cerdo ibérico Manchado de Jabugo (MJ) tiene su origen a finales del siglo XIX en el término municipal de dicha población onubense con la participación entre otros, de cerdos ibéricos rubios y negros de la zona. En la actualidad se encuentra catalogada en peligro de extinción (Forero, 1999). Tradicionalmente está vinculada al área de dehesa del suroeste de la península ibérica, aunque con expansión en los últimos años a otras zonas de España. Es un animal rústico que aprovecha muy bien las montaneras y que es capaz de proporcionar paletas y jamones de gran aceptación.
El programa de conservación del Manchado de Jabugo sigue abierto y, de forma paralela, se han ido incorporando otros para mejoras parámetros como el retrocruzamiento que trata de superar la fuerte consanguinidad que existía como consecuencia de la escasez de ejemplares.
Sin embargo, su situación censal actúa hizo que la Diputación de Huelva y la Universidad de Córdoba firmaran el proyecto titulado “Puesta en marcha del Programa de Recuperación por Retrocruzamiento y Conservación de la variedad de Cerdo Ibérico Manchado de Jabugo”. En este proyecto se plantearon dos acciones específicas: una cuyo objetivo es la conservación de la variedad del cerdo Ibérico Manchado de Jabugo en pureza y otra la recuperación de esta variedad tan amenazada mediante retrocruzamientos.
Un retrocruzamiento se refiere al cruce de un descendiente hibrido de primera generación con uno de los padres o con un genotipo idéntico al paterno, en este caso sería de animales puros de Manchado de Jabugo y animales de la estirpe Retino línea Villalón (Vill).
Esta estirpe tiene su origen a principios del siglo XX a partir del cruce entre verracos retintos portugueses y hembras retintas extremeñas, surge así el denominado “retinto mejorado” del cual se originará de manera directa, por selección en núcleo cerrado, la línea Villalón (Clemente y cols., 2006).
El objetivo de este estudio fue utilizar los retrocruzamientos con la estirpe Villalón, una estirpe filogenéticamente próxima para incrementar los tamaños reales y efectivos del Manchado de Jabugo y a través del uso de marcadores moleculares hacer un seguimiento de la integración de los animales retro-absorbidos en la población original.
MATERIAL Y MÉTODOS
Muestreo
Se analizaron 93 muestras de pelo de animales nacidos entre el año 2013 y 2017, y pertenecientes a las fincas “Las Peñasquillas” y “Huerto Ramírez”. La extracción del DNA se ha realizado con el método Kawasaki (1990). Un panel de 25 microsatélites específicos de la especie porcina fueron establecidos de acuerdo a las recomendaciones de la FAO y de la Sociedad Internacional de Genética Animal (ISAG). Se analizaron mediante la técnica de la reacción en cadena de la polimerasa (PCR) y la separación por tamaños de los fragmentos obtenidos se ha realizado mediante una electroforesis en gel de poliacrilamida en un secuenciador automático ABI 377XL (Applied Biosystems, Foster City, CA, USA). El análisis de los fragmentos y la tipificación alélica se ha realizado mediante los programas informáticos Genescan Analysis 3.1.2 y Genotyper 2.5, respectivamente.
Análisis estadístico
Se calcularon la Heterocigosidad esperada y observada y el número de alelos mediante la extensión MS Tools del programa EXCEL (Park, 2001). Se comparan los resultados con los encontrados en otras líneas/variedades del Ibérico: Villalón, Dorado gaditano, Retinto, Entrepelado, Torbiscal, Lampiño y Manchado de Jabugo. Los animales de Manchado de Jabugo analizados en este proyecto se incorporan a la población referencia de Manchado de Jabugo de la base de datos del Grupo de Investigación PAI-AGR-218 del Departamento de Genética de la Universidad de Córdoba. Además, se incluyeron razas comerciales como son: Duroc, Pietrain, Large White y Landrace. Se calcularon las distancias genéticas DA de Nei (Nei y cols., 1983) y se construye un árbol de distancias con el programa Populations (Langella, 1999). El árbol se dibuja mediante la utilización del programa Treeview (Page, 1996).
La asignación individual de cada animal a una raza se realiza utilizando el programa Structure (Pritchard y cols., 2000), que se basa en un método no supervisado que asume una situación de equilibrio de Hardy-Weinberg para las frecuencias de los alelos, estimando para cada individuo incluido en el análisis la probabilidad de que pertenezca a cada una de las poblaciones consideradas ancestrales. Esta probabilidad es en realidad la distribución posterior de cada porcentaje de genoma que proviene de las poblaciones ancestrales y es calculada aplicando un enfoque bayesiano utilizando técnicas MCMC (Monte Carlo Markow Chain). Este procedimiento presenta como ventajas más destacables el no requerir especificar las frecuencias alélicas de las poblaciones ancestrales y el permitir tener en cuenta situaciones genéticas complejas, incluyendo el caso de muestras que provienen de mezcla entre varias poblaciones. Para realizar este estudio se utilizará la base de datos del Laboratorio de Genética Molecular Aplicada que contiene todas las posibles poblaciones ancestrales que podrían haber contribuido en la conformación del genoma de los animales estudiados. Se realiza un análisis en los que se tienen en cuenta las siguientes líneas/variedades del Ibérico: Retinto del Andévalo, Villalón, Dorado gaditano, Retinto, Entrepelado, Torbiscal, Lampiño y Manchado de Jabugo. Se introducen también otras razas porcinas como Duroc, Pietrain, Large White, Landrace y un cruce comercial Landrace X Large White.
Retrocruzamiento
Como se muestra en la figura 1, se utilizaron los retrocruzamientos con la línea Villalón, estirpe filogenéticamente próxima al Manchado de Jabugo para incrementar el tamaño real y efectivo de la población.

Si los cruzamientos se realizar según los resultados obtenidos en la asignación individual, la progresión teórica/esperada de la absorción por retrocruzamiento en el pedigrí sería como se muestra en la tabla 1.
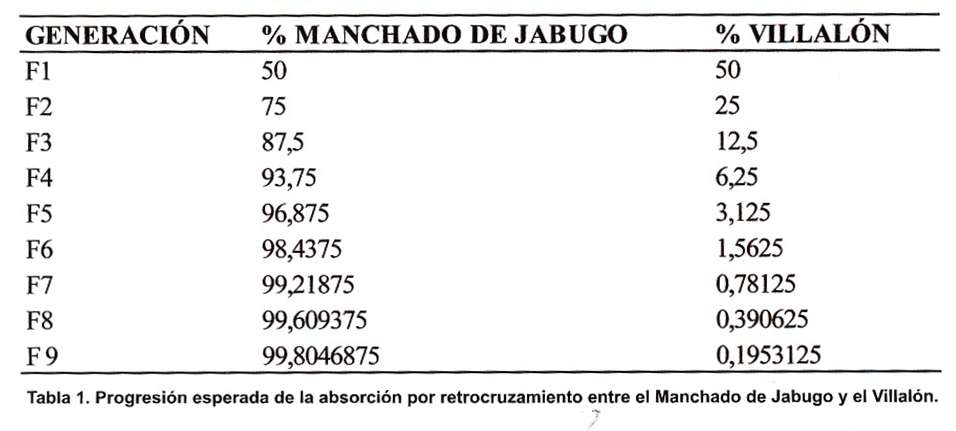
RESULTADOS Y DISCUSIÓN
La Diputación de Huelva junto al Grupo PAIDI AGR218 de la Universidad de Córdoba vienen trabajando conjuntamente en la recuperación de una variedad tan importante en el sector porcino como es el Manchado de Jabugo. Uno de los objetivos perseguidos fue la mejora de la cantidad y calidad de información genealógica que será empleada posteriormente en la planificación de los apareamientos dirigidos. Desde la firma del proyecto de investigación, transcurridos ya 5 años, cada anualidad se ha ido recopilando toda la información genealógico generada y los valores de asignación individual por los ejemplares analizados, teniendo así información que ha permitido planificar de forma objetiva los cruzamientos entre las 2 variedades próximas genéticamente.
Un pedigrí bien estructurado y la inclusión permanente de los animales más próximos a la línea pura MJ garantizaran la supervivencia de la raza. En la figura 2 se observa la evolución de la información molecular respecto a los coeficientes de asignación individual se refiere. Esta tendencia positiva es resultado de la aplicación correcta de los resultados en campo por parte de los ganaderos, produciéndose un éxito para la conservación de esta variedad tan emblemática del cerdo ibérico, como es el Manchado de Jabugo.
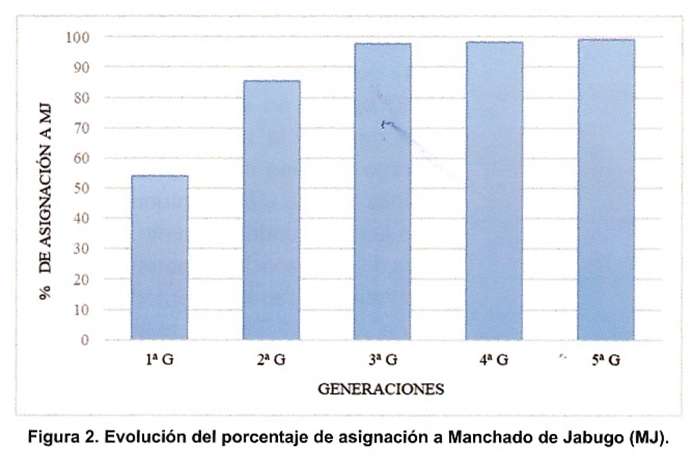
Por otro lado, no debemos olvidar el mantenimiento de la diversidad genética de la población, por ello cada año se analizaba y evaluaba estadísticamente los resultados moleculares obtenidos. En la tabla 2 se muestran los principales indicadores de la diversidad genética en las distintas poblaciones incluidas en el estudio.
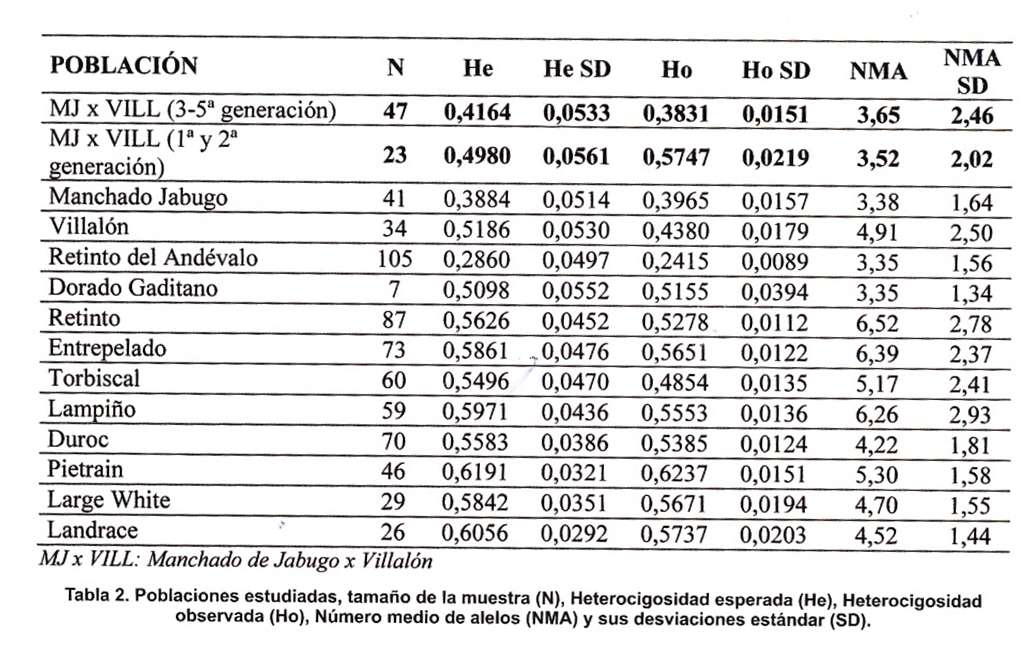
Para los 25 microsatélites analizados, un promedio de 4,66 alelos son detectados en los 707 individuos de las 14 poblaciones estudiadas, un valor más bajo (5,14) del observado por Gama y cols., (2013) aunque en el estudio se incluyen razas de toda la península ibérica, sin embargo, en línea con lo encontrado por Martínez y cols., (2000) en un estudio con todas las variedades del ibérico. La media de la heterocigosidades esperada y observada en la estirpe MJ x Vill fueron de 0,457±0,05 y 0,479±0,02, respectivamente. Mientras que para el resto de las poblaciones fue de 0,530±0,04 y 0,502±0,02, respectivamente.
La variabilidad genética en los animales de la estirpe MJ x Vill de la 3ª a la 5ª generación es inferior a la encontrada en la línea pura Villalón (0,438), con niveles de Heterocigosidad observada inferiores (0,383). Sin embargo, son superiores a los hallados en el Manchado de Jabugo puro de la población de referencia. Con respecto al resto de las variedades y razas incluidas en el estudio, esta estirpe presenta valores inferiores a las demás líneas/variedades de cerdo ibérico introducidas en el estudio, a excepción del Retinto del Andévalo, que presentó una HO DE 0,242. Por otro lado, haciendo una comparativa entre los resultados observados en la estirpe MJ x Vill se aprecia un descenso de los niveles de variabilidad de los animales a partir de la 3ª generación con respecto a los animales de la 1ª y 2ª generación. Aunque los valores de variabilidad genética como el número de alelos y la heterocigosidad observada son bajos, hay que considerar que se están comparando variedades de una raza que por el solo efecto de muestreo aportan un valor bajo, pero siempre en línea con lo observado en otras razas locales de cerdos. El hecho que el retro cruce vuelva a valores bajos de estos parámetros era de esperarse en cuanto se va sustituyendo el genoma de la raza.
En la figura 3 se observan las relaciones genéticas entre las poblaciones estudiadas, en ella se identifican dos clústeres. El primer clúster observado corresponde al grupo de las razas del tronco ibérico que comparten un origen común y su distribución geográficamente cerca, mientras en el otro clúster se ubica las razas comerciales. Dentro del grupo de los ibéricos en este dendrograma se observa como la estirpe MJ x Vill ocupa una posición muy próxima a la línea pura de Manchado de Jabugo, alejándose de la otra línea de la que procede (Villalón).
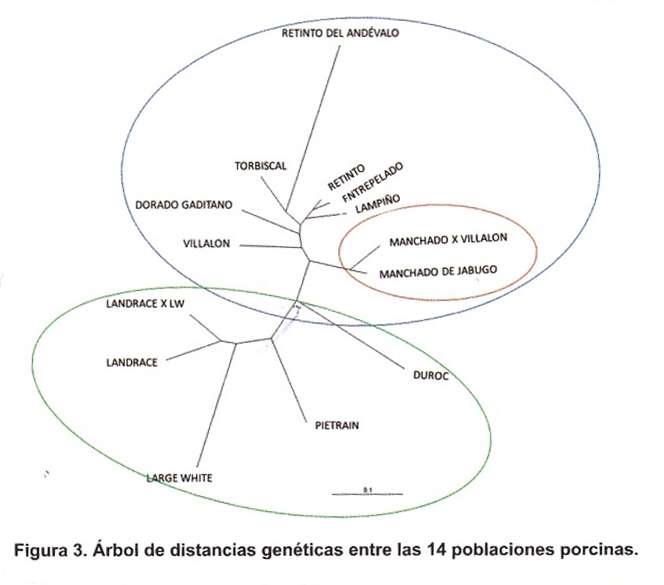
Como era de esperarse, el Manchado de Jabugo y su cruce con la variedad Villalón se sitúan en la misma rama del árbol a una distancia genética muy baja, pero el dato más importante que refleja este análisis es que todos los individuos utilizados convergen dentro del agrupamiento de las variedades ibéricas subrayando su pureza genética, entendida como ausencia de cruce con animales de razas mejorantes (Duroc, etc).
El enfoque Bayesiano implementado por el programa Structure, fue usado para estimar el número más probable de poblaciones subyacentes a la diversidad genética observada (Gama y cols., 2013). El número de poblaciones ancestrales inferidas fueron K=2 hasta el K=15, con un total de 10 repeticiones para cada valor de K. Utilizando la prueba de Evano y cols., (2005) se determinó que el K=6 es el número más probable de poblaciones ancestrales que contribuyen a la variabilidad genética observada en las 14 poblaciones observadas (figura 4). Los animales de la estirpe MJ x Vill se agrupan junto a los Manchados de Jabugo puros y solo algunos animales de la 1ª y 2ª generación muestran signos de los cruces de las variedades Manchado de Jabugo (en naranja) y Villalón (en amarillo), aunque estos individuos muestran diversas proporciones de ambas líneas (tabla 3).
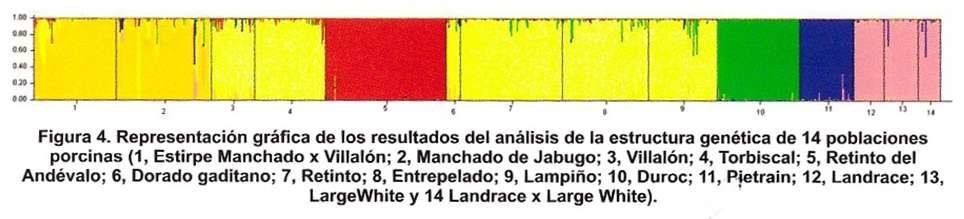
Por otro lado, algunos animales están compuestos por líneas de color verde (Duroc) y azul (Pietrain). Esto puede deberse a la presencia de animales que contienen un porcentaje de genoma de estas razas ocasionado probablemente a un cruce lejano. Por lo tanto, esta herramienta no solo nos permite monitorear la composición genómica del retrocruce, sino también a detectar individuos cuya “pureza” genética obliga a una utilización más atenta en el plan de conservación del MJ.
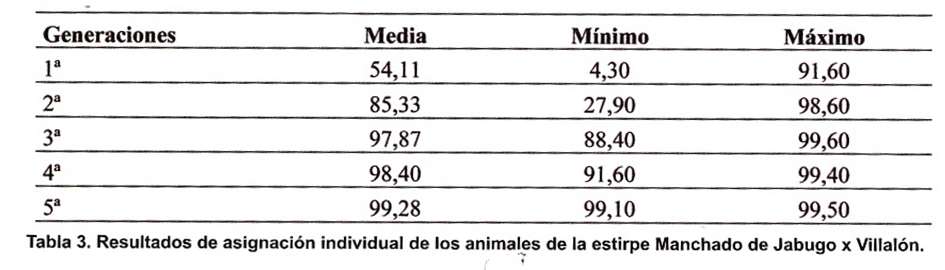
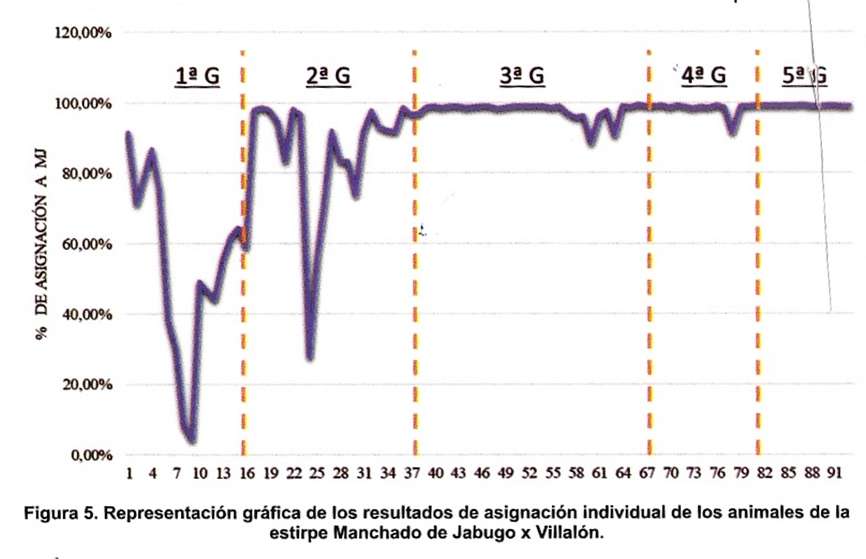
Los valores de asignación individual expresados en porcentaje, de cada uno de los animales analizados, se encuentra en la tabla 3. Los animales de la 1ª generación presentan valores muy dispares, de manera que algunos se asignan a la variedad Manchado de Jabugo con valores elevados (91,60%), mientras que otros presentan valores muy bajos (4,30%). En la 2ª generación, los valores son en general más elevados, aunque sigue habiendo animales con porcentajes de asignación reducidos. Sin embargo, los animales de la 3ª generación de retrocruzamientos se asignan a Manchado de Jabugo, con valores similares a los presentados por los animales Manchado de Jabugo puros. Lo mismo sucede en los animales de la 4ª generación: genéticamente pueden considerarse animales Manchado de Jabugo puros.
CONCLUSIONES
Estos resultados muestran que los animales Manchado de Jabugo x Villalón sometidos a retrocruces con Manchado de Jabugo se podrían integrar en la población pura de Manchado de Jabugo a partir de la 3ª generación. Con el desarrollo de este proyecto se ha conseguido aumentar el tamaño real y efectivo de la población, garantizando su supervivencia y minimizando su erosión genética.
Autores:
- Forero J., y Venegas M. Diputación de Huelva.
- Gómez M.M., Landi V., Martínez A.M., y Delgado J.V. Departamento de Genética, Facultad de Veterinaria. Universidad de Córdoba.


Deja un comentario