Resumen
Las razas porcinas ibérica y alentejana se encuentran biogeográficamente localizadas en el suroeste de la península Ibérica. Estas razas comparten un conjunto de características comunes. Ambas son fenotípicamente muy similares; muestran un bajo índice de crecimiento y una elevada deposición grasa y tienen un sistema común de producción al aire libre en el que se explotan los recursos de la Dehesa. El objetivo del presente trabajo, dentro del marco del proyecto TREASURE, consistió en caracterizar la diversidad genética de ambas razas usando los datos de genotipado de 39 polimorfismos localizados en 34 genes previamente asociados a distintos caracteres de interés económico. Estos polimorfismos fueron genotipados en 950 cerdos pertenecientes a las razas ibérica y alentejana y a 18 razas europeas autóctonas adicionales. Los bajos valores para heterozigosidad observada y esperada indican una baja diversidad genética en ibéricos y alentejanos. Las distancias genéticas estimadas usando DS y FST fueron cercanas a 0 (0,007 y 0,039, respectivamente), sugieren que estas razas son genéticamente muy similares. Asimismo, las técnicas de análisis multivariante utilizadas como análisis de componentes principales y asignación a grupos mostraron que los individuos de estas razas están muy próximos formando clústeres diferenciados del resto de razas. Estos resultados concuerdan con los de otros autores que ya mostraron distancias genéticas cortas entre ambas razas usando secuencias de ADN mitocondrial.
INTRODUCCIÓN
La ibérica y la alentejana son razas de cerdo mediterráneas que tienen un origen genético común y están localizadas en el suroeste de la península Ibérica. Los cerdos alentejanos están localizados principalmente en Portugal y la población de cerdos ibéricos se encuentra principalmente en España. Aunque ha existido intercambio de individuos entre ganaderos de ambos países, principalmente hembras reproductoras y verracos, este intercambio se ha visto limitado por un efecto frontera.
Los cerdos ibéricos y los alentejanos tienen características comunes, ambas razas tienen un crecimiento muy inferior comparado con otras razas comerciales de cerdo blanco como duroc o landrace y tienen una elevada deposición grasa, lo cual está relacionado con una elevada calidad de sus productos derivados. Además, ambas razas tienen un sistema de producción extensivo, en el cual los animales se crían al aire libre y tienen una alimentación basada principalmente en hierba y bellota. Las carnes provenientes de los cerdos de ambas razas son de una elevada calidad y son la base de la producción de productos locales (Silió, 2000; Ramos et al., 2003).
Sin embargo, también tienen características diferenciales. Como es bien sabido, un gran porcentaje de la producción ibérica está basada en cruces de al menos del 50% con duroc, ya que los individuos cruzados tienen un mayor porcentaje de ganancia media diaria que los ibéricos puros (Pérez-Serrano, 2008; Sánchez-Esquiliche, 2011). Por otro lado, aunque en el sistema de producción de alentejano no existe una estrategia de cruces con otras razas tan asentada como en ibérico, sí que se han datado cruces con berkshire y landrace durante los años 50 (Frazão, 1984).
El proyecto multidisciplinar TREASURE tiene como objetivo principal caracterizar la diversidad de razas locales y sistemas de producción para productos sostenibles y de alta calidad. Uno de los principales objetivos del proyecto consiste en la caracterización de la diversidad genética de una serie de razas locales, incluidas la alentejana y la ibérica, mediante el uso de variantes genéticas/polimorfismos que son variaciones en la secuencia del ADN entre los individuos de una población (Figura 1). Para llevar a cabo esta caracterización, se genotiparon variantes genéticas en individuos ibéricos, alentejanos y en otras 18 razas autóctonas de porcino.
MATERIALES Y MÉTODOS
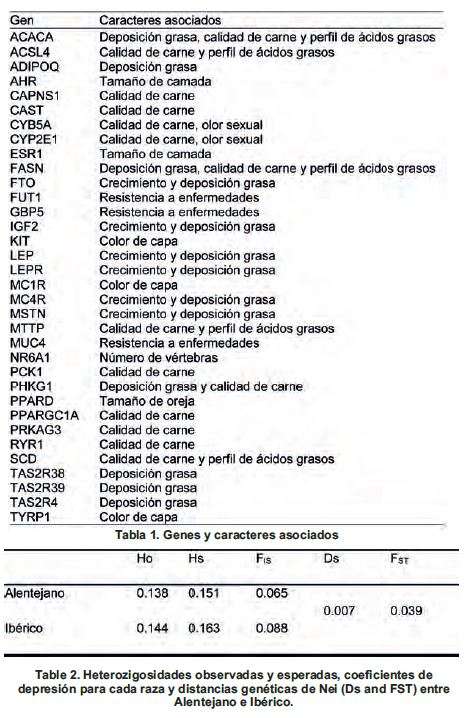
Se incluyó en el estudio un total de 96 animales alentejanos e ibéricos (48 de cada raza) y 854 animales de otras 18 razas autóctonas (Apulo Calabrese, Basque, Bísaro, Black Majorcan, Black Sicilian, Black Slavonian, Casertana, Cinta Senese, Gascon, Krskopolje, Lithuanian indigenous wattle, Mangalitsa, Mora Romagnola, Moravka, Old Lithuanian White, Sarda, Schwäbisch Hällisches, Turopolje). El ADN se extrajo de muestras de sangre periférica con un protocoloestándar de fenol-cloroformo (Sambrook et al., 1989). Después de realizar una priorización con los genes candidatos más interesantes relacionados con caracteres productivos, de calidad, de resistencia a enfermedades, morfológicos y reproductivos, se seleccionó un panel de 39 polimorfismos localizados en genes candidatos y mutaciones causales (Tabla 1) para genotiparlos mediante la plataforma OpenArray™ (Thermo Fisher Scientific, Figura 2).

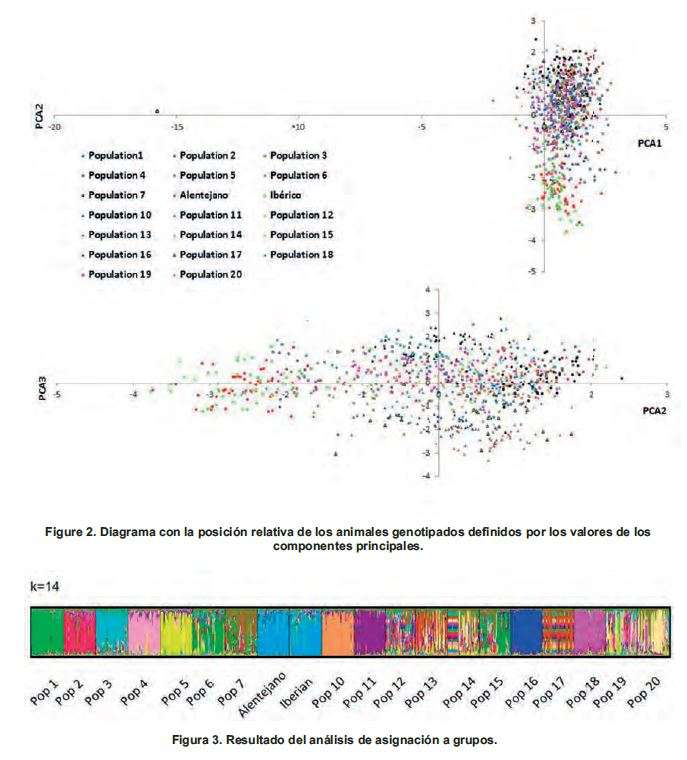
A partir de los datos de genotipado de los polimorfismos, se calcularon medidas de variabilidad genética como la heterocigosidad observada (Ho) y esperada (He) así como el coeficiente de consanguinidad (Fis) (Nei, 1987). Además, se estimaron las distancias genéticas Ds y Fst, basadas en las frecuencias alélicas de los polimorfismos estudiados en las diferentes poblaciones, de acuerdo con Takezaki y Nei (1996). Asimismo, se realizó un análisis de componentes principales para representar las relaciones geométricas entre las diferentes razas (Lê et al., 2008). Por último, se realizó un análisis de asignación a grupos utilizando el programa STRUCTURE.
RESULTADOS Y DISCUSIÓN
El 95% de las muestras fueron genotipadas con éxito. Las frecuencias alélicas de los polimorfismos genotipados en los individuos ibéricos y alentejanos fueron similares. Los valores promedio de Ho y Hs por polimorfismo están comprendidos entre 0,024 y 0,414 y 0,025 u 0,415, respectivamente. El valor promedio de Fst es igual a 0.26, lo que indica que un 26% de las diferencias observadas son debidas a diferencias entre razas, y el 74% restante, son debidas a diferencias entre individuos. En 14 de los polimorfismos genotipados, uno de los dos alelos está fijado, y los 25 restantes están segregando en ambas poblaciones a frecuencias alélicas similares. Además, en ambas razas los valores de heterozigosidad (Ho y Hs) son parejos y bajos, al igual que los valores de FIS, lo que sugiere una diversidad genética baja en las variantes genéticas estudiadas. Asimismo, las distancias genéticas (Ds y Fst) fueron también pequeñas, lo cual sugiere que las razas analizadas son genéticamente muy similares (Tabla 1).
En la Figura 2 aparecen representados el primer componente principal frente al segundo (Figura 2a) y el segundo frente al tercero (Figura 2b). En esta figura, los puntos verdes representan a los individuos ibéricos y los rojos, a los alentejanos y tanto en una representación como en la otra se observa que ambas razas aparecen agrupadas y juntas dentro del mismo clúster y separadas del resto de razas autóctonas europeas.
Estos resultados se vieron corroborados con el análisis realizado utilizando el software STRUCTURE, en el cual, los individuos de las razas alentejana e ibérica eran asignados al mismo grupo (Figura 3).
Van Asch et al. (2012) ya estimaron distancias genéticas cortas entre ibéricos y alentejanos cuando analizaron la región control del ADN mitocondrial. Estos mismos autores también dieron cuenta de un intenso flujo de genes entre las poblaciones de jabalí e ibérico y alentejano de las zonas analizadas.
Teniendo en cuenta los resultados obtenidos en el presente estudio, los cuales muestran una cercanía genética muy grande en las variantes genéticas analizadas, podría deducirse que las razas ibéricas y alentejana fueran estirpes pertenecientes a una misma raza, más que razas independientes por sí misma. Dentro del proyecto TREASURE se están realizando análisis utilizando un mayor número de información genética a lo largo de todo el genoma para corroborar los resultados mostrados en el presente estudio. Estos resultados, junto con la información para el resto de las poblaciones será presentada en un futuro.
El proyecto TREASURE está financiado por la Unión Europea en el marco del programa Horizonte 2020 (proyecto número 63477). El contenido de este artículo refleja únicamente la visión de los autores y la agencia ejecutiva de investigación no se hace responsable del uso que pueda hacerse de la información contenida.
Autores
- Centro de I+D en Cerdo Ibérico, Dpto. Mejora Genética Animal, INIA. María Muñoz, Juan M. García-Casco.
- Department of Agrifood Production and Environmental Science – University of Firenze (Italy). Riccardo Bozzi, Alessandro Crovetti.
- ICAAM – Instituto de Ciências Agrárias e Ambientais Mediterrânicas, Universidade de Évora, Évora (Portugal). Rui Charneca, José M. Martins.
- INIA, Departamento de Mejora Genética Animal, Madrid (Spain). María Muñoz, Ana I. Fernández, Juan M. García-Casco, Cristina Óvilo.
- Department of Agricultural and Food Sciences, University of Bologna, Bologna (Italy). Luca Fontanesi.


Deja un comentario