ORIGEN Y EVOLUCIÓN DEL PRRSV
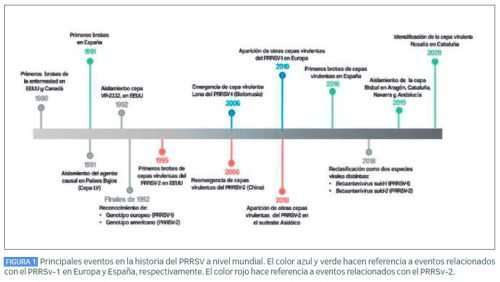
A finales de la década de 1980, se describe una nueva enfermedad en diferentes granjas de porcino en EE.UU. y Canadá asociada a unos brotes con fallos respiratorios y reproductivos. Debido a que se desconocía el agente causal, esta enfermedad recibió diferentes nombres como “enfermedad misteriosa porcina” o “síndrome respiratorio y de infertilidad porcina” (denominado como SIRS, del inglés “Swine Infertility and Respiratory Syndrome”). Al mismo tiempo, aparecieron diferentes brotes asociados a signos clínicos similares en varias granjas de Europa, que fueron descritos con diferentes nombres como “Abortus blauw” en Holanda, “Blue ear pig disease” en el Reino Unido o “Porcine Epidemic Abortion and Respiratory Syndrome” (PEARS) en Alemania.
El virus, agente causal de esta enfermedad, se aisló en Lelystad (Países Bajos), en 1991, proponiéndose como nombre de la enfermedad el acrónimo “PEARS”, ya que, con este acrónimo, que contenía la palabra “ears”, orejas en inglés, se hacía referencia al enrojecimiento de las orejas, una lesión típica de los lechones infectados en ese momento, aunque no hubo consenso al respecto del uso de esta denominación.
En EE.UU., en 1992, un año después, se aisló una 2ª cepa del mismo virus, denominada VR-2332. Cepas que desde su aislamiento son consideradas las cepas de referencia para cada territorio: la cepa Lelystad (LV) para Europa y la cepa VR-2332 para Norteamérica.
A finales de 1992, durante el 1º Simposio Internacional sobre este síndrome, hasta ese momento conocido como SIRS en EE.UU. y Canadá, la enfermedad recibió por consenso su nombre definitivo: Síndrome Reproductivo y Respiratorio Porcino (PRRS, del inglés “Porcine Reproductive and Respiratory Syndrome”), denominándose virus del PRRS (PRRSV), al agente etiológico que lo producía. En la Figura 1 se representan los eventos más importantes en la historia del PRRSV.
El análisis genético posterior de las cepas de referencia (LV y VR-2332) reveló que tan sólo compartían el 55-70% de su secuencia de nucleótidos. Este hallazgo, junto con la observación de una enorme variabilidad antigénica entre ambas cepas, llevó a considerar dos genotipos biológica y genéticamente diferentes del virus, designados como genotipo europeo o tipo 1 (PRRSV-1) y genotipo americano o tipo 2 (PRRSV-2), siendo LV la cepa de referencia para el PRRSV-1 y la VR-2332 para el PRRSV-2. Posteriormente se han identificado a su vez 4 subtipos filogenéticos, subtipos 1-4, en el PRRSV-1; mientras que para el PRRSV-2, han sido definidos 9 linajes diferentes.
Con el paso del tiempo, la identificación de un mayor número de diferencias genéticas entre ambos genotipos ha llevado a que, recientemente, se hayan reclasificado ambos genotipos del virus como 2 especies virales distintas, Betaarterivirus suid-1 (PRRSV-1) y Betaarterivirus suid-2 (PRRSV-2), dentro del género Betaarterivirus (familia Arteviridae, orden Nidovirales).
EMERGENCIA Y REEMERGENCIA DE LAS CEPAS VIRULENTAS DEL PRRSV
En 1995 se describe en el suroeste de Iowa (EE.UU.) el 1º brote producido por una cepa virulenta del PRRSV. Este brote estuvo asociado a la cepa VR-2385, que se aisló en una granja con un elevado número de abortos tardíos y numerosos casos de enfermedad respiratoria grave, con una alta mortalidad tanto en lechones como en cerdos en crecimiento. Entre 1996 y 1997, se aislaron, en granjas próximas geográficamente, otras cepas virulentas como la SDSU 73, la JA 142 y la 17198-6, que se asociaron a brotes con alta mortalidad de cerdas y “tormentas” de abortos. A finales de 2001, también en EE.UU., concretamente en Minnesota, se identificaron numerosos brotes producidos por cepas virulentas de PRRSV, conocidos como “aislado MN 184”, que provocaban una alta mortalidad en cerdas y enfermedad respiratoria grave en cerdos en crecimiento. La Figura 2 muestra los principales brotes causados por cepas virulentas en el mundo.
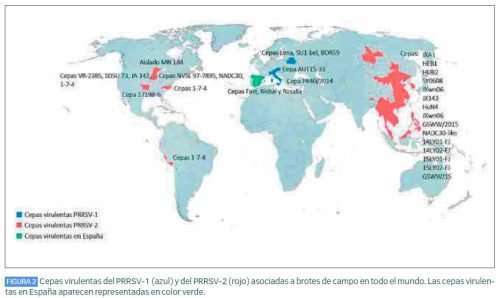
Tras 5 años de latencia, las cepas virulentas de PRRSv reemergieron en el sureste asiático. Así, en el verano de 2006, se notificó por 1ª vez en China un brote de una enfermedad denominada inicialmente como “enfermedad de la fiebre alta porcina” (“high fever disease”), que afectó a más de 2.000.000 de cerdos en 10 provincias diferentes. La enfermedad se caracterizaba por producir fiebre alta (40-42°C), petequias y equimosis multifocales, y una elevada tasa de mortalidad en las explotaciones. Ante la clínica y lesiones que se observaron en estos brotes, diferentes enfermedades como la Peste Porcina Africana, la Peste Porcina Clásica, el PRRSV o la septicemia provocada por bacterias como Streptococcus spp. y Mycoplasma spp. fueron incluidas en el diagnóstico diferencial de este brote. Finalmente, los ensayos moleculares y experimentos posteriores demostraron que diferentes cepas virulentas, pertenecientes al PRRSV-2, como JXA1, JX143 y HuN4, entre otras, eran las causantes de la epidemia.
Estas cepas virulentas de PRRSV-2, comenzaron a extenderse rápidamente por el continente asiático, afectando a diferentes países, como Vietnam, Bután, Camboya, Laos, Malasia, Myanmar, Filipinas, Tailandia y Singapur, llegando también a regiones de Corea del Sur y Rusia. Desde entonces, también se han notificado numerosos brotes causados por la recombinación de estas mismas cepas en diferentes estados de los EE.UU., como los brotes provocados por la cepa 1-4-4, así como en países sudamericanos como Perú.
Coincidiendo con la aparición del 1º brote en China, en Europa se aisló, en una granja bielorrusa, una cepa del PRRSv denominada Lena, que producía fiebre alta, problemas reproductivos, debilidad en los neonatos, así como graves trastornos respiratorios y una alta mortalidad en cerdos en crecimiento. La evaluación de los hallazgos clínicos y las lesiones observadas en este brote reveló similitudes con las cepas virulentas americanas aisladas en la década de 1990. Sin embargo, el análisis filogenético reveló que la cepa Lena era muy diferente de la cepa de referencia VR-2332 del PRRSV-2, resultando finalmente ser una cepa virulenta del PRRSV-1 (subtipo 3). 4 años más tarde, en 2010, se aisló en Bielorrusia otra cepa virulenta del PRRSV-1, también del subtipo 3, denominada SU1-bel, que se caracterizaba por una marcada hipertermia y una enfermedad clínica grave, acompañada de mayores lesiones pulmonares en comparación con las cepas de campo.
Inicialmente, la virulencia de las cepas de PRRSV-1 fue asociada con el subtipo 3, como las cepas Lena y SU1-bel; sin embargo, diferentes cepas de los subtipos 1 y 2, como la BOR59 (subtipo 2), la PR40/2014 (subtipo 1) y la AUT15-33 (subtipo 1) han sido asociadas con brotes de alta mortalidad de PRRSV-1 en Bielorrusia, Italia y Austria, respectivamente. Estas cepas originan graves fallos reproductivos en madres e intensos signos de enfermedad respiratoria, causando una alta mortalidad en la fase de destete, no sólo en las granjas libres de PRRSV sino también en las vacunadas.
APARICIÓN DE LAS CEPAS VIRULENTAS EN ESPAÑA
Desde las primeras descripciones de la presencia del virus del PRRS en España, hacia el año 1991, han sido descritas varias cepas virulentas en nuestro país. La mayoría de las veces la información de su diseminación fue limitada, probablemente, por una mayor capacidad de contención de estas cepas a nivel de granja o región, sea por las características de las cepas implicadas o por la información disponible durante esos años. La instauración de programas de control regional de PRRS en España en la zona de Tauste, en el norte de la provincia de Zaragoza, hace ya más de 10 años, permitió emplear un sistema de detección y comunicación de brotes que, junto con la secuenciación de las cepas implicadas, permitió tipificar en 2016 una de las primeras cepas virulentas, principalmente diseminada por granjas de Aragón y Cataluña, denominada “Fast” (rápida en inglés) (Figura 1).
Un año más tarde, se detectaron varios brotes producidos por cepas virulentas en granjas de Castilla y León, en concreto en las provincias de Segovia y Burgos. La iniciación de un plan de control regional del PRRS en la provincia de Segovia ese mismo año permitió tipificar, una vez más, una cepa que causaba estos signos clínicos, sobre todo a nivel reproductivo y respiratorio en la fase de transición y cebo, no habituales en los casos clínicos ocasionados y diagnosticados anteriormente por los veterinarios de la zona. Se detectaron casos únicamente en las zonas anteriormente mencionadas hasta 2019, cuando derivó genéticamente a otras cepas y/o variantes menos virulentas.
Posteriormente, en el año 2019, se detectó una cepa en distintas granjas de Aragón, Cataluña, Navarra y Andalucía con un probable nexo epidemiológico común, que se identificó como la cepa Bisbal. Se caracterizaba por una clínica reproductiva atípica y una vez más, problemas respiratorios e infecciones secundarias en la fase de transición. Una de las particularidades de esta cepa fue la dificultad de estabilizar las granjas donde se diagnosticó, por los largos períodos de detección de su presencia en lechones al nacimiento, y posteriormente en las fases de lactación y transición. Actualmente, se continúa detectando de forma minoritaria en algunas granjas de las mismas comunidades autónomas dónde se describió por 1ª vez.
A principios de 2020, en el noreste de Cataluña, se detectaron varios brotes virulentos con mortalidades altas, incluso en cerdas reproductoras. Las cargas virales elevadas, los períodos de excreción mayores y la morbilidad de animales de todas las edades combinado con los movimientos de éstos a granjas de otras zonas del país, fueron probablemente algunos de los factores que permitieron tipificarla como la cepa virulenta con más diseminación descrita en España hasta la fecha, siendo identificada como la cepa Rosalía. Desde las primeras secuenciaciones, se pudo comprobar que las cepas más cercanas filogenéticamente a la cepa Rosalía fueron detectadas previamente en granjas de Italia. Desde 2021, un porcentaje elevado de las variantes de la cepa Rosalía descritas en el campo no ocasionan los brotes virulentos que se describieron en 2020. Durante la descripción de los diferentes brotes ocasionados por las cepas virulentas de PRRSV, tanto a nivel mundial como en España, se emplearon diferentes términos para denominarlas: “cepas atípicas”, “cepas virulentas” o “cepas patogénicas”, entre otros. Es por ello que, recientemente, con el objetivo de homogenizar la terminología, se ha propuesto el término “cepa virulenta” como el término más correcto para hacer referencia a estas cepas. Además, se ha sugerido una 1ª aproximación de las principales características que debe cumplir una cepa para ser considerada como virulenta (Figura 3).
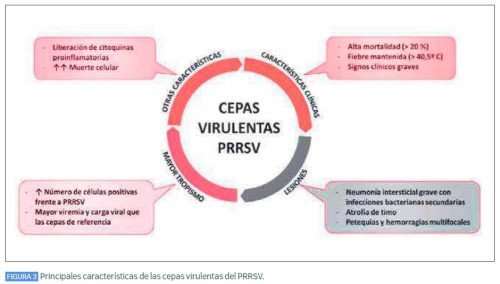
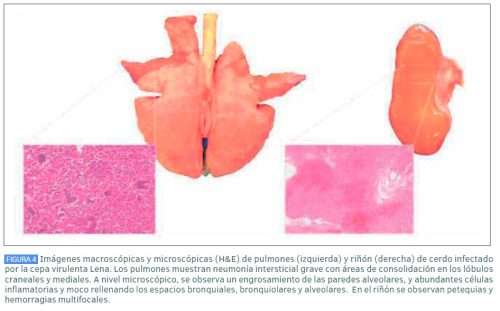
Las cepas virulentas de PRRSV dan lugar a una clínica muy aguda, provocando una elevada hipertermia en los animales (40,5-42ºC) y un aumento de la morbilidad y mortalidad (20% o mayor) en las granjas, afectándose varios grupos de edad. Entre las lesiones más llamativas provocadas por estas cepas, destaca una neumonía intersticial grave generalmente acompañada por una bronconeumonía supurativa o fibrinosa, que indicaría la participación de bacterias oportunistas (Figura 4). Además, es común encontrar una marcada atrofia de timo, linfadenopatías, y petequias y hemorragias multifocales en diferentes órganos como riñón (Figura 4), corazón, nódulos linfáticos y corazón.
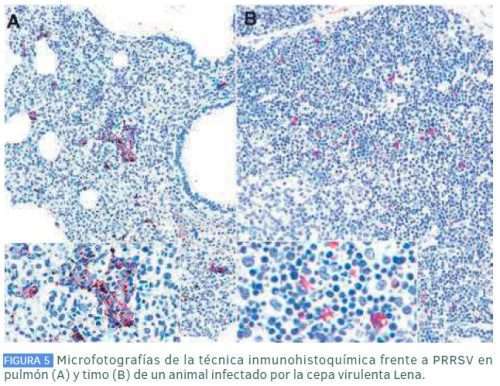
Las cepas de PRRSV provocan una viremia más alta que las cepas clásicas del virus, que se acompaña de un mayor número de células positivas frente a PRRSV (Figura 5) y cargas virales elevadas y mantenidas en pulmón y en órganos linfoides como el timo, la tonsila y los nódulos linfáticos. Estudios comparativos han demostrado que estas cepas presentan una mayor capacidad para inducir mecanismos de muerte celular en las células del sistema inmune en comparación con las cepas de campo, deteriorando los mecanismos de defensa del hospedador. Este hecho ayudaría a explicar la gravedad de los signos clínicos y lesiones observadas en los cerdos afectados.
La aparición de nuevas cepas virulentas del PRRSV ha suscitado una enorme preocupación en la industria porcina y en la comunidad investigadora, debido a la importación de animales y a la falta de vacunas plenamente protectoras. Además, se debe prestar especial atención a la posible recombinación entre las cepas endémicas y las emergentes, que puede dar lugar a un escenario favorable para la aparición de nuevos brotes devastadores, y pueden confundirse clínica y lesionalmente con otras enfermedades como la Peste Porcina Africana, como sucedió con la aparición de los brotes virulentos en el continente asiático.
Fuente: Anaporc


Deja un comentario